自己紹介¶
辻 真吾(つじしんご)
東京大学先端科学技術研究センター ゲノムサイエンス分野(そろそろ任期切れ)
Pythonスタートブック(技術評論社)
機械学習に関してはユーザ−。手法自体の研究者ではありません。
30分で全体を理解すると言ってしまったので、
とにかく詰め込みます!¶
機械学習のオープンなツール群
An overview of free software tools for general data mining, 2014, DOI:10.1109/MIPRO.2014.6859735
R¶
○マニアックな統計関数が充実
×汎用言語ではない(個人的にはRの文法が馴染めない)
IDEを使うと楽。RStudioがお薦め https://www.rstudio.com/
http://www.r-bloggers.com/top-6-reasons-you-need-to-be-using-rstudio/
ここはPython勉強会なので、やっぱり
scikit-learn¶
http://scikit-learn.org/stable/
Pythonの機械学習ライブラリ。機能充実。GUIは無いので、すこしわかりにくい?
導入は、毎度のことながら、Anacondaがお薦め
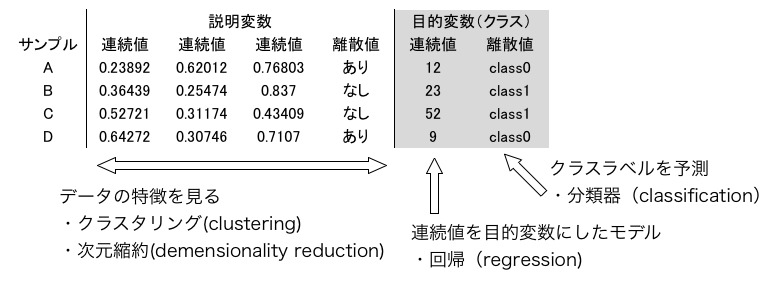
機械学習のおおまかな分類¶
PCA¶
Principal Component Analysis(主成分分析)
高次元データの次元を減らす。サンプルが最もばらついている方向から新たな軸を設定(第1主成分、第2主成分・・・)
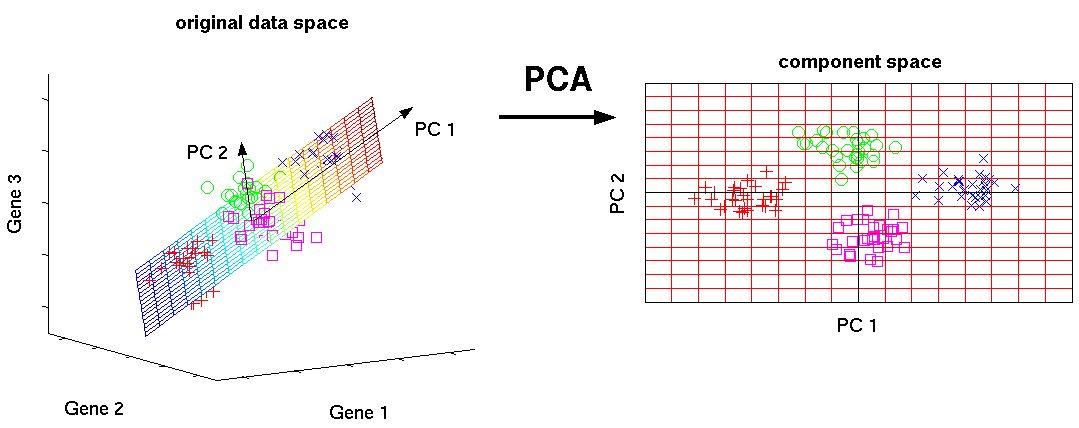 http://phdthesis-bioinformatics-maxplanckinstitute-molecularplantphys.matthias-scholz.de/
http://phdthesis-bioinformatics-maxplanckinstitute-molecularplantphys.matthias-scholz.de/
# サンプルデータの生成
# 200サンプル、20変数、2クラス
from sklearn.datasets.samples_generator import make_classification
X,y = make_classification(n_samples=200, n_features=20, n_informative=2, n_redundant=0, n_repeated=0, n_classes=2)
print('20の変数を持った200サンプル')
print(X.shape)
print('0か1。どちらのクラスに属するか')
print(y.shape)
20の変数を持った200サンプル (200, 20) 0か1。どちらのクラスに属するか (200,)
# 20次元はプロットできないので、2変数を選んでプロット
# 参考までに色を付けてあります。
%matplotlib inline
import pylab
pylab.figure(figsize=(8,6))
for a,b,_y in zip(X[:,2],X[:,6],y):
c = 'b' if _y else 'r'
pylab.scatter(a,b,c=c)
# PCAを使って2次元に次元を縮約
from sklearn.decomposition import PCA
pca = PCA(n_components=2)
pca_X = pca.fit_transform(X)
pca_X.shape
(200, 2)
# 第1主成分と第2主成分をプロット
pylab.figure(figsize=(8,6))
for x,_y in zip(pca_X,y):
c = 'b' if _y else 'r'
pylab.scatter(x[0],x[1],c=c)
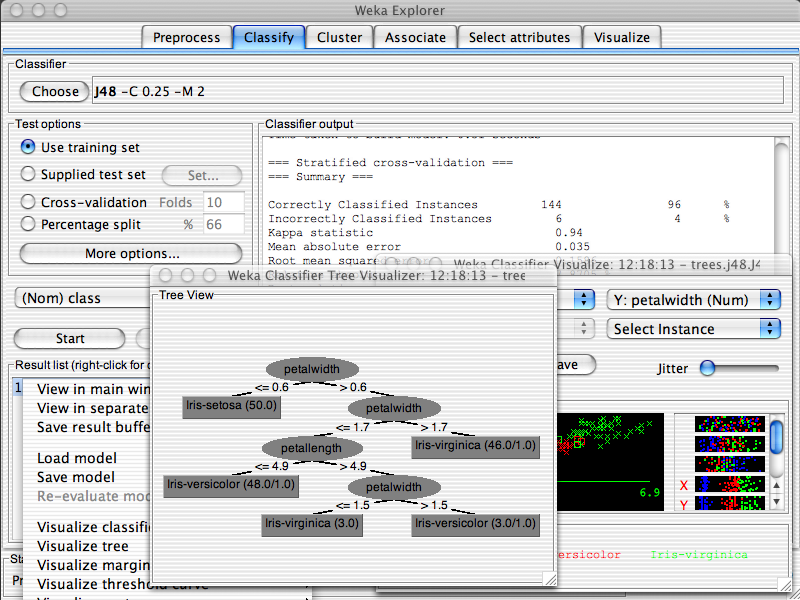
K-meansクラスタリング¶
もっともシンプルなクラスタリング手法の1つ。ランダムな開始点から始めて、近くのサンプルをクラスター化。その後クラスの重心に新たな中心を設定。
from sklearn.cluster import KMeans
kmeans = KMeans(n_clusters=2)
pred_y = kmeans.fit_predict(X)
print(pred_y)
# 答えは見てない。データの特徴だけを使って、サンプルを分類している
[0 1 0 1 0 0 1 1 0 0 1 0 1 1 0 1 1 0 1 1 0 1 0 0 1 1 0 0 1 1 1 1 1 1 1 0 0 0 1 0 0 0 1 1 1 1 1 1 0 0 0 1 0 0 1 1 0 0 1 1 0 0 0 1 0 0 1 1 0 0 1 0 0 0 1 1 0 1 0 1 1 1 1 0 1 1 1 1 1 1 1 1 0 0 1 0 1 0 0 0 0 1 1 0 0 0 1 0 1 0 0 0 1 1 0 0 0 0 0 0 1 0 1 1 0 1 0 1 0 0 0 0 1 0 1 0 0 1 0 0 1 0 0 0 0 1 1 0 1 0 1 0 0 0 0 1 1 1 0 0 1 1 1 1 0 1 1 0 0 1 0 0 1 0 0 1 0 0 0 1 0 1 1 0 1 0 0 1 1 1 0 0 1 0 0 1 0 1 0 1]
# PCAのプロットに、K-meansの結果を重ねる
pylab.figure(figsize=(8,6))
for x,_y in zip(pca_X,pred_y):
c = 'b' if _y else 'r'
pylab.scatter(x[0],x[1],c=c)
他にもいろいろ。K-means以外は、パラメータの設定が面倒な印象
scikit-learnに実装されているクラスタリング手法の一覧
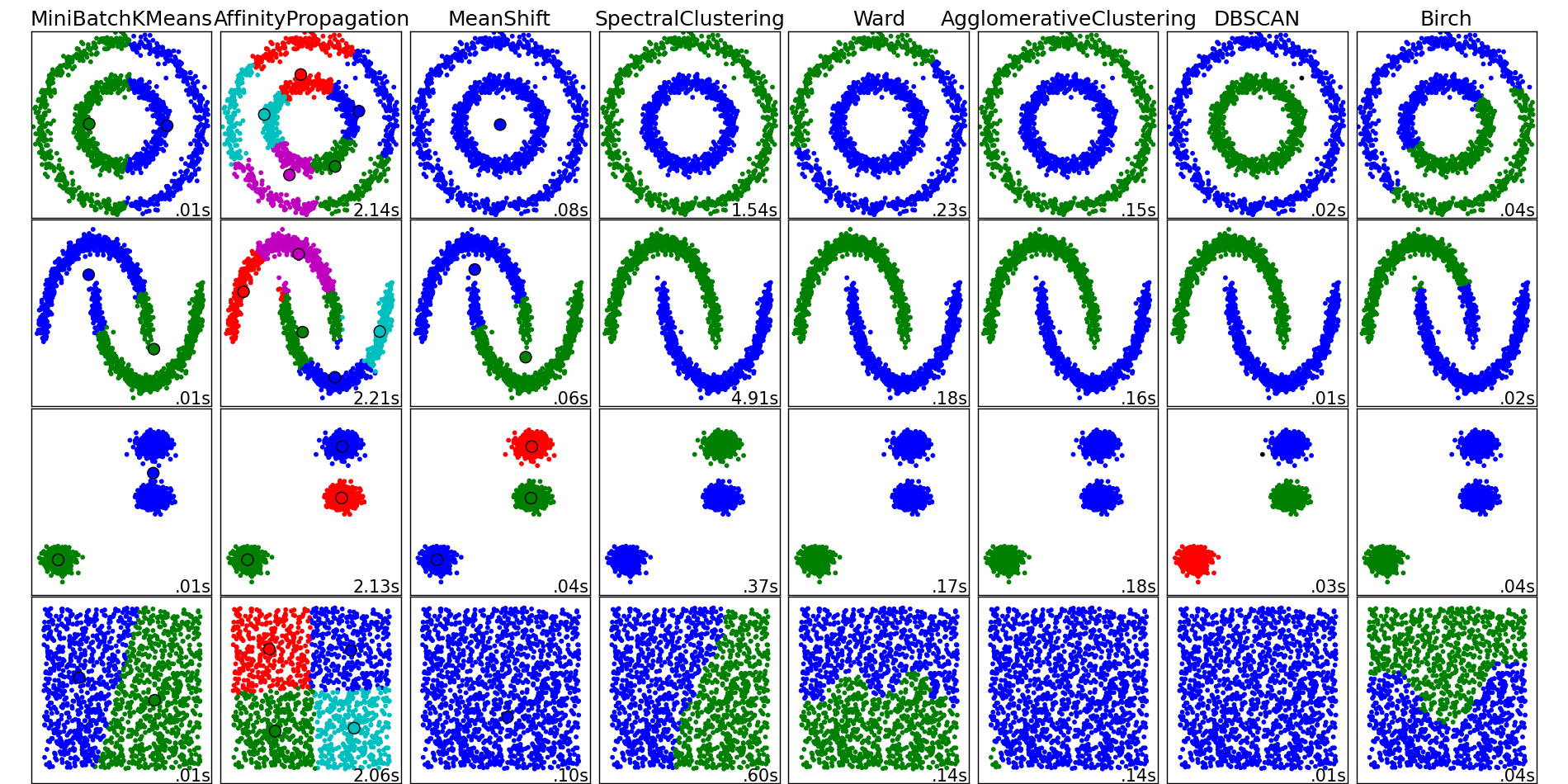 http://scikit-learn.org/stable/auto_examples/cluster/plot_cluster_comparison.html
http://scikit-learn.org/stable/auto_examples/cluster/plot_cluster_comparison.html
予測モデルの構築¶
教師あり学習。元々のサンプルの分類を使って、新たなサンプルのクラスを予測する
- SVM(Support Vector Machine)
- 決定木
- Random Forests
- Deep Learning
などなど
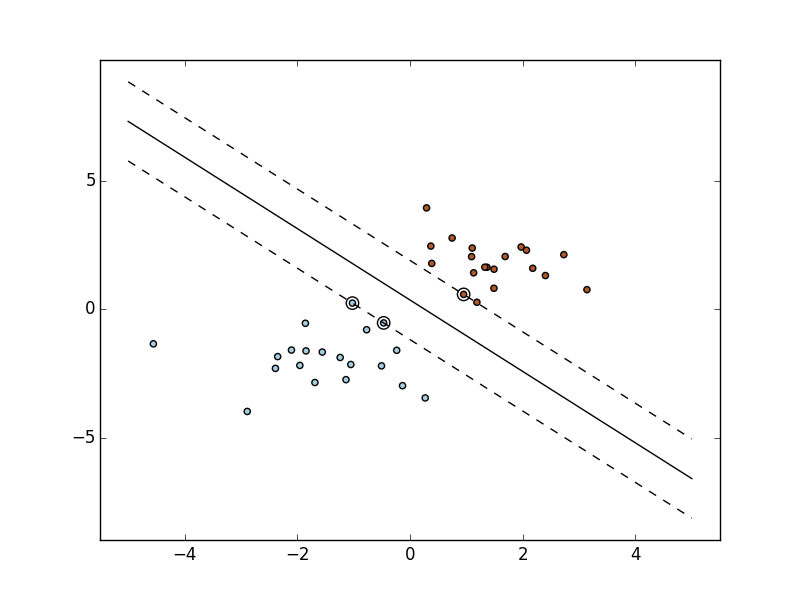
SVM(Support Vector Machine)¶
Kernel trick¶
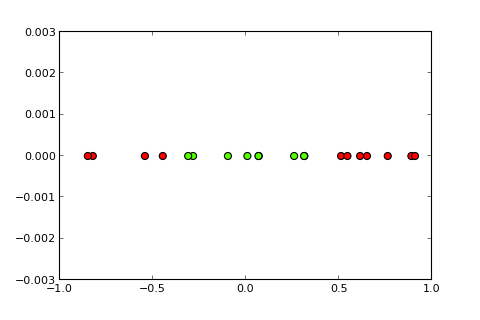 xをx2に変換
xをx2に変換
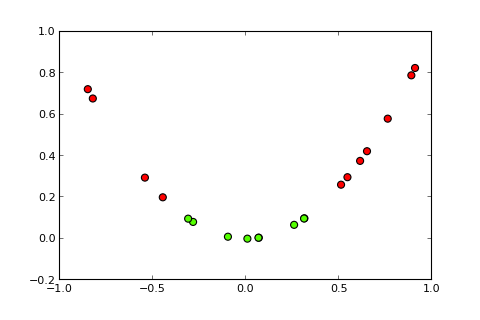 http://peekaboo-vision.blogspot.jp/2012/12/kernel-approximations-for-efficient.html
http://peekaboo-vision.blogspot.jp/2012/12/kernel-approximations-for-efficient.html
from sklearn.svm import SVC
clf = SVC()
clf.fit(X, y)
y_pred = clf.predict(X)
# もとのデータを予測しても2個ほど間違える。
from sklearn.metrics import accuracy_score
accuracy_score(y, y_pred)
# モデルを作るのに使ったデータを予測しても仕方無いので・・・
0.98999999999999999
# 仮想的にテストサンプルを作る
import numpy as np
import random
# 0-199の配列から、20個ランダムに選ぶ
test_idx = random.sample(range(200), 20)
# 長さ200の全部Trueが入ったnumpy.arrayを用意
mask = np.ones(X.shape[0],dtype=bool)
# テストサンプルに使うところだけを、Falseに
mask[test_idx] = False
test_X = X[test_idx]
train_X = X[mask]
test_y = y[test_idx]
train_y = y[mask]
print(test_X.shape)
print(train_X.shape)
(20, 20) (180, 20)
clf = SVC()
clf.fit(train_X, train_y)
# モデルの作成に使っていないサンプルのクラスを予測
y_pred = clf.predict(test_X)
accuracy_score(test_y, y_pred)
# 20個中1個外す
0.94999999999999996
# Leave One Out Cross Validation (LOOCV)
# サンプル全体から1つ取り出して、残りでモデルを作って、とっておいたサンプルを予測
# その他、さまざまなCross Validationの方法がある
from sklearn import cross_validation
hit_cnt = 0
for train_idx, test_idx in cross_validation.LeaveOneOut(200):
clf = SVC()
clf.fit(X[train_idx], y[train_idx])
if y[test_idx] == clf.predict(X[test_idx])[0]: hit_cnt+=1
print('{}/200'.format(hit_cnt))
196/200
パラメータの設定と性能評価¶
デフォルトを使っていましたが、SVCにCとgammaというパラメータがある。
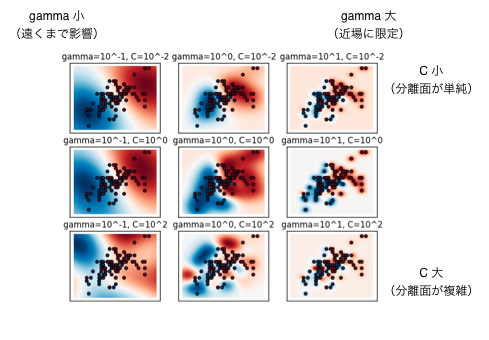
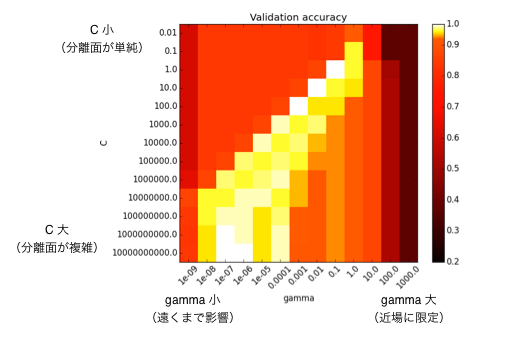 http://scikit-learn.org/stable/auto_examples/svm/plot_rbf_parameters.html
http://scikit-learn.org/stable/auto_examples/svm/plot_rbf_parameters.html
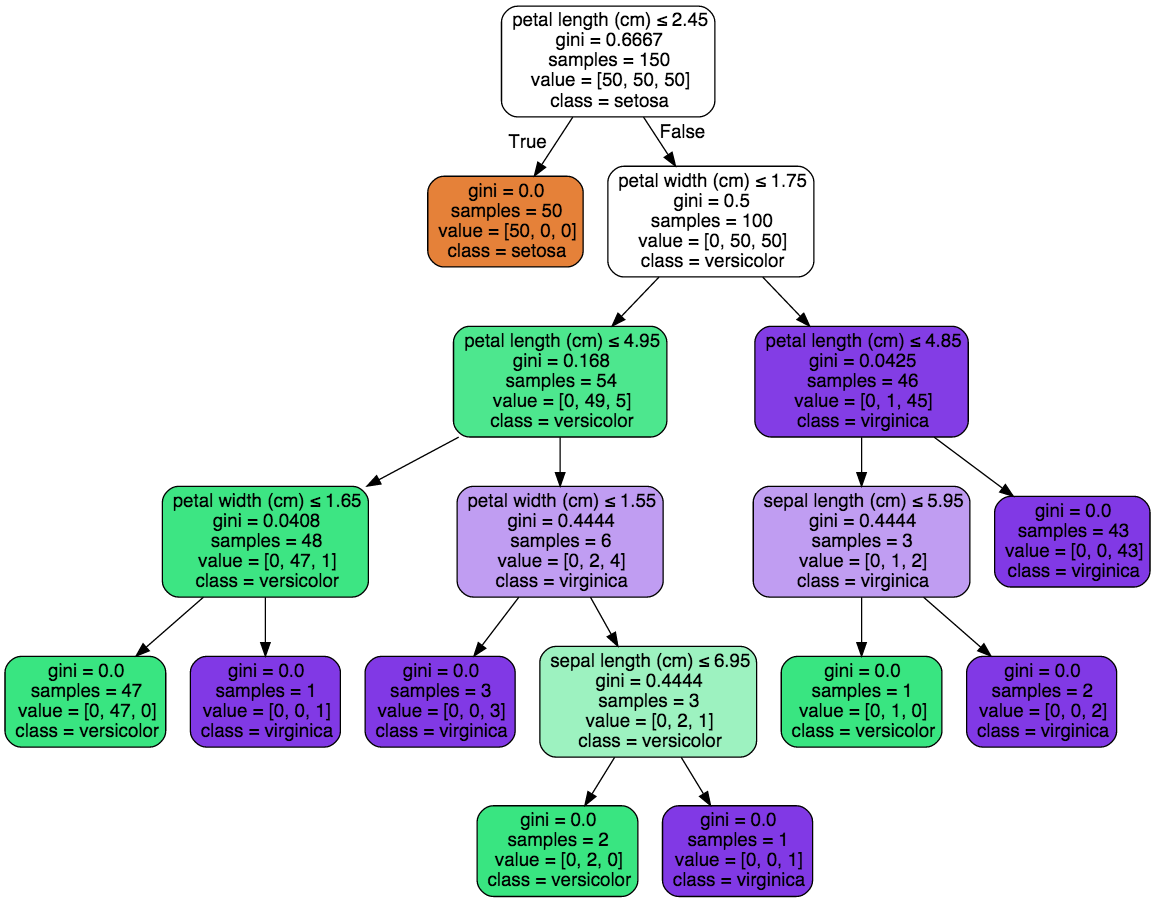
決定木(Decision Tree)¶
結果が可視化しやすいので、医療の診断など、80年代の人工知能ブームでエキスパートシステムなどに利用される。
Random Forests¶
「手元のサンプルからランダムに選び出したサンプルで決定木を作る」という作業を繰り返す方法。木がたくさんあるので森。
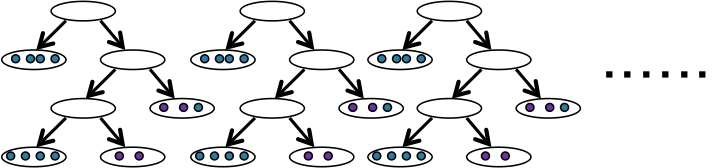
興味深い側面がいくつかあります
- large P small N問題への対応:サンプル(N)が少なく、変数(P)が多いと多くの機械学習アルゴリズムの性能が低下することへの対応
- アンサンブルメソッド:復習の学習アルゴリズムを使って、予測の精度を上げる方法
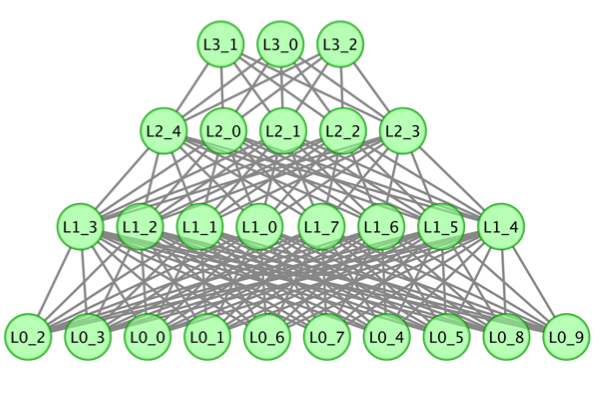
Deep Learning¶
Google Brainと呼ばれる大規模なコンピュータの上で稼働するDeep Neural Networkが教師信号なしに、1,000万枚の画像データから、人や猫の顔を学習することに成功
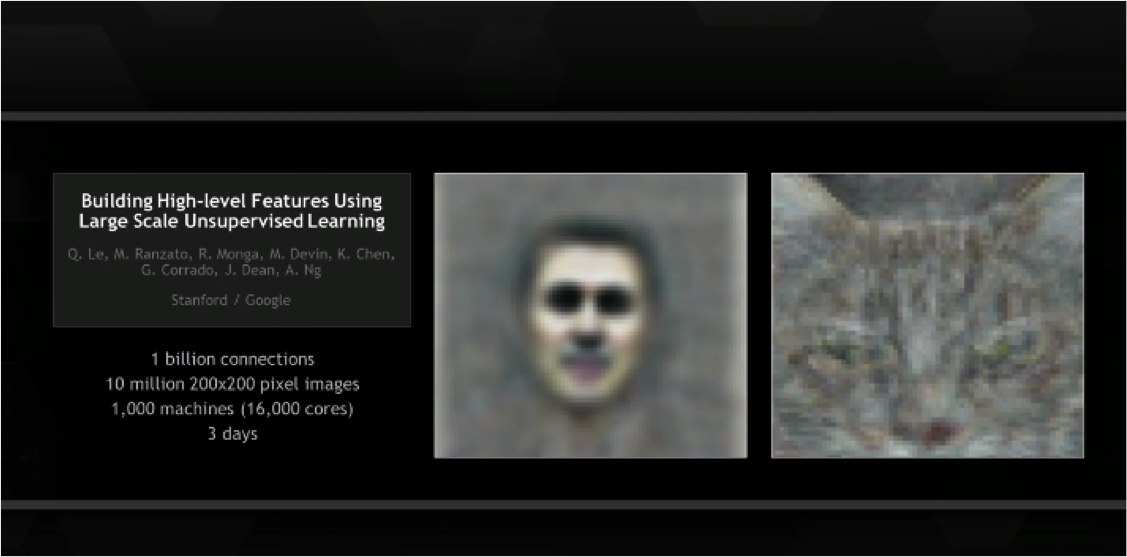
Keynote presentation of Jen-Hsun Huang, Co-Founder and CEO, NVIDIA GPU Technology Conference (GTC) 2014
Deep Learningの中身は、多層のニューラルネットワーク
これ自体の発想は、昔からあった。
近年、層ごとの事前の学習最適化(Greedy Layer-Wise Pre-Training)が、性能向上に大きく貢献することが発見された。
ジェフリー・ヒントン

実際に使ってみた¶
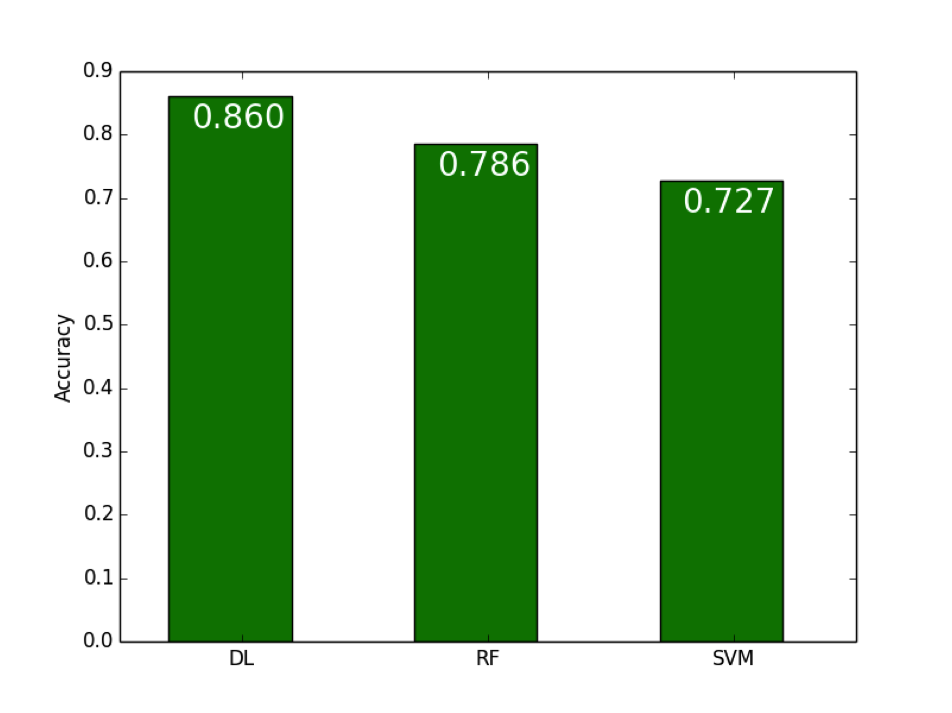
がんの細胞からとられたデータで、そのがんの種類を当てるモデル。単純な分類器としてもよい性能。
積み残した話題¶
- 回帰分析から一般化線型モデルへ続くどちらかというと統計的な話
- 過学習(overfitting) 手元のサンプルばかりを性能良く予測できるモデルを作ってしまい、新しいサンプルへの予測性能が落ちる現象(汎化性能の低下)
変数の選択(Recursive feature eliminationなど)
実際のデータへの応用
参考資料¶
- 機械学習によるデータ分析まわりのお話専門用語も多いですが、非常に良くまとまっている。
- 深層学習
 Deep Learningの日本語情報としては、最高にわかりやすい。
Deep Learningの日本語情報としては、最高にわかりやすい。